samtools和picard的排序问题
samtools和picard都有对SAM/BAM文件进行排序的功能,一般都是基于坐标排序(还提供了-n选项来设定用reads名进行排序),先是对chromosome/contig进行排序,再在chromosome/contig内部基于start site从小到大排序,对start site排序很好理解,可是对chromosome/contig排序的时候是基于什么标准呢?
基于你提供的ref.fa文件中的chromosome/contig的顺序。当你使用比对工具将fastq文件中的reads比对上参考基因组后会生成SAM文件,SAM文件包含头信息,其中有以@SQ开头的头信息记录,reference中有多少条chromosome/contig就会有多少条这样的记录,而且它们的顺序与ref.fa是一致的。
SAM/BAM文件的头信息:
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
> @SQ SN:chr1 LN:195471971
> @SQ SN:chr2 LN:182113224
> @SQ SN:chr3 LN:160039680
> @SQ SN:chr4 LN:156508116
> @SQ SN:chr5 LN:151834684
> @SQ SN:chr6 LN:149736546
> @SQ SN:chr7 LN:145441459
> @SQ SN:chr8 LN:129401213
> @SQ SN:chr9 LN:124595110
> @SQ SN:chr10 LN:130694993
> @SQ SN:chr11 LN:122082543
> @SQ SN:chr12 LN:120129022
> @SQ SN:chr13 LN:120421639
> @SQ SN:chr14 LN:124902244
> @SQ SN:chr15 LN:104043685
> @SQ SN:chr16 LN:98207768
> @SQ SN:chr17 LN:94987271
> @SQ SN:chr18 LN:90702639
> @SQ SN:chr19 LN:61431566
> @SQ SN:chrX LN:171031299
> @SQ SN:chrY LN:91744698
> @SQ SN:chrM LN:16299
> @RG ID:ERR144849 LB:ERR144849 SM:A_J PL:ILLUMINA
>
ref.fa中chromosome/contig的排列顺序:
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
> >chr2
> >chr3
> >chr4
> >chr5
> >chr6
> >chr7
> >chr8
> >chr9
> >chr10
> >chr11
> >chr12
> >chr13
> >chr14
> >chr15
> >chr16
> >chr17
> >chr18
> >chr19
> >chrX
> >chrY
> >chrM
>
它们的顺序一致
当使用samtools或picard对SAM/BAM文件进行排序时,这些工具就会读取头信息,按照头信息指定的顺序来排chromosome/contig。所以进行排序时需要提供包含头信息的SAM/BAM文件。
那么普通情况下我们的chromosome/contig排序情况是什么样的?
一般情况下我们获取参考基因组序列文件的来源有三个:
- NCBI
- ENSEMBEL
- UCSC Genome Browser
这里以UCSC FTP下载源为例:
这是一个压缩文件,使用
tar zxvf chromFa.tar.gz解压后,会得到多个fasta文件,每条chromosome/contig一个fasta文件:chr1.fa, chr2.fa …之后我们会将它们用
cat *.fa >ref.fa合并成一个包含多条chromosome/contig的物种参考基因组序列文件用
grep ">" ref.fa可以查看合并后发ref.fa文件中染色体的排列顺序为:
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
> >chr11
> >chr12
> >chr13
> >chr14
> >chr15
> >chr16
> >chr17
> >chr18
> >chr19
> >chr1
> >chr1_GL456210_random
> >chr1_GL456211_random
> >chr1_GL456212_random
> >chr1_GL456213_random
> >chr1_GL456221_random
> >chr2
> >chr3
> >chr4
>
这和我们平时想象的染色体的排列顺序是不是有一些出入?难道不应该是从chr1开始到chr22,最后是chrX和chrY这样的顺序吗?
想象归想象,实际上它是按照字符顺序进行的,chr11就应该排在chr2前面
一般情况下在进行SAM文件的排序时,染色体的排序到底是按照哪种规则进行排序的,不是一个很重要的问题,也不会对后续的分析产生影响,但是在执行GATK流程时,GATK对染色体的排序是有要求的,必须按照从chr1开始到chr22,最后是chrX和chrY这样的顺序,否则会报错
面对这样变态的要求,我们怎么解决?
在构造ref.fa文件时,让它按照从chr1开始到chr22,最后是chrX和chrY这样的顺序进行组织就可以了:
1 | for i in $(seq 1 22) X Y M; |
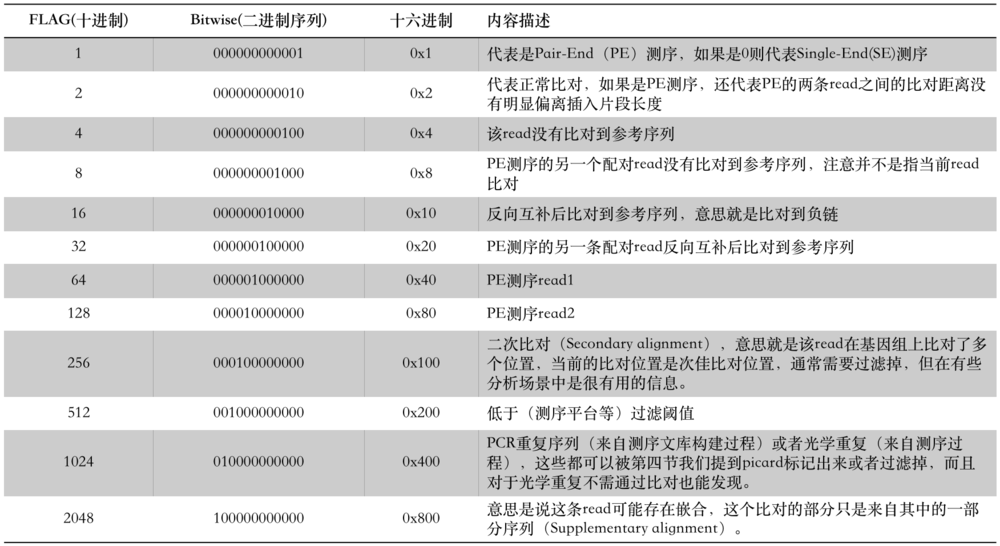
SAM文件中FLAG值的理解
FLAG列在SAM文件的第二列,这是一个很重要的列,包含了很多mapping过程中的有用信息,但很多初学者在学习SAM文件格式的介绍时,遇到FLAG列的说明,常常会一头雾水

what?还二进制,这也太反人类的设计了吧!
不过如果你站在开发者的角度去思考这个问题,就会豁然开朗
在mapping过程中,我们想记录一条read的mapping的信息包括:
- 这条read是read1 (forward-read) 还是read2 (reverse-read)?
- 这条read比对上了吗?与它对应的另一头read比对上了吗?
- …
这些信息总结起来总共包括以下12项:
| 序号 | 简写 | 说明 |
|---|---|---|
| 1 | PAIRED | paired-end (or multiple-segment) sequencing technology |
| 2 | PROPER_PAIR | each segment properly aligned according to the aligner |
| 3 | UNMAP | segment unmapped |
| 4 | MUNMAP | next segment in the template unmapped |
| 5 | REVERSE | SEQ is reverse complemented |
| 6 | MREVERSE | SEQ of the next segment in the template is reversed |
| 7 | READ1 | the first segment in the template |
| 8 | READ2 | the last segment in the template |
| 9 | SECONDARY | secondary alignment |
| 10 | QCFAIL | not passing quality controls |
| 11 | DUP | PCR or optical duplicate |
| 12 | SUPPLEMENTARY | supplementary alignment |
而每一项又只有两种情况,是或否,那么我可以用一个12位的二进制数来记录所有的信息,每一位表示某一项的情况,这就是原始FLAG信息的由来,但是二进制数适合给计算机看,不适合人看,需要转换成对应的十进制数,也就有了我们在SAM文件中看到的FLAG值
但是FLAG值所包含信息的解读还是要转换为12位的二进制数
SAM文件中那些未比对的reads
SAM格式文件的第3和第7列,可以用来判断某条reads是否比对成功到了基因组的染色体,左右两条reads是否比对到同一条染色体

有两个方法可以提取未比对成功的测序数据:
- SAM文件的第3列是*的(如果是PE数据,需要考虑第3,7列)
2
>
- 或者SAM文件的flag标签包含0x4的
2
3
> $ samtools view -f4 sample.bam sample.unmapped.sam
>
虽然上面两个方法得到的结果是一模一样的,但是这个perl脚本运行速度远远比不上上面的samtools自带的参数
对于PE数据,在未比对成功的测序数据可以分成3类:
- 仅reads1没有比对成功
该提取条件包括:
- 该read是read1,对应于二进制FLAG的第7位,该位取1,其十进制值为64;
- 该read未成功比对到参考基因组,对应于二进制FLAG的第3位,该位取1,其十进制值为4;
- 另一配对read成功比对到参考基因组,对应于二进制FLAG的第4位,该位取0,其十进制值为8;
2
3
4
> # 对于取0的位点采取过滤的策略,用-F参数,值设为8
> $ samtools view -u -f 68 -F 8 alignments.bam >read1_unmap.bam
>
- 仅reads2没有比对成功
该提取条件包括:
- 该read是read2,对应于二进制FLAG的第8位,该位取1,其十进制值为128;
- 该read未成功比对到参考基因组,对应于二进制FLAG的第3位,该位取1,其十进制值为4;
- 另一配对read成功比对到参考基因组,对应于二进制FLAG的第4位,该位取0,其十进制值为8;
2
3
4
> # 对于取0的位点采取过滤的策略,用-F参数,值设为8
> $ samtools view -u -f 132 -F 8 alignments.bam >read2_unmap.bam
>
- 两端reads都没有比对成功
该提取条件包括:
- 该read未成功比对到参考基因组,对应于二进制FLAG的第3位,该位取1,其十进制值为4;
- 另一配对read未成功比对到参考基因组,对应于二进制FLAG的第4位,该位取1,其十进制值为8;
2
3
> $ samtools view -u -f 12 alignments.bam >pairs_unmap.bam
>
看完这一部分,是不是有一个感觉:FLAG玩得溜,SAM文件可以处理得出神入化
为什么一条read会有多条比对记录?
首先,思考一个问题:对于PE数据,一条测序片段(fragment)有read1和read2两条测序片段,它们俩的名字相同,那么对于这一条测序片段,对它进行mapping之后得到的SAM文件中会出现几条记录呢?
先声明以下只对BWA比对得到的SAM文件进行讨论,对其他比对工具输出的SAM文件可能不适用
首先,基于经验积累告诉我,它会得到有且只有两条记录,原因在于,BWA在对每条read执行比对时只会给出一个hit,若这条read是multiple mapping的情况,它会从中选择MAPQ值最高的那个hit作为输出,若存在多个hit的MAPQ值相等且最高,那么BWA会从中随机选择一个作为输出
对于我的这个假设可以用以下的方法进行验证:
1 | # 将SAM文件的第一列提出来,排序去重,同时统计每个QNAME出现的次数 |
上面的测试结果与我们的假设吻合
但是在一次处理三代测序数据(三代测序数据是Single-End)中发现了不同:
1 |
在输出中出现了一些不太和谐的结果:有极少部分的QNAME对应2条以上的记录,这意味着存在一条read会有多条比对记录的情况,why?
对这个与预期不完全相符的结果,尝试去寻找里面的原因,其间进行了各种各样的推理、假设、验证,最终在李恒的github中找到了答案
2. Why does a read appear multiple times in the output SAM?
BWA-SW and BWA-MEM perform local alignments. If there is a translocation, a gene fusion or a long deletion, a read bridging the break point may have two hits, occupying two lines in the SAM output. With the default setting of BWA-MEM, one and only one line is primary and is soft clipped; other lines are tagged with 0x800 SAM flag (supplementary alignment) and are hard clipped.
参考资料: